入门教程¶
主要讲述VMD中常见的图形界面(鼠标)操作。
VMD软件界面介绍¶
注解
VMD的稳定版本是1.9.3,本教程以1.9.3版本进行介绍。 大部分内容也适用于其他版本的VMD。
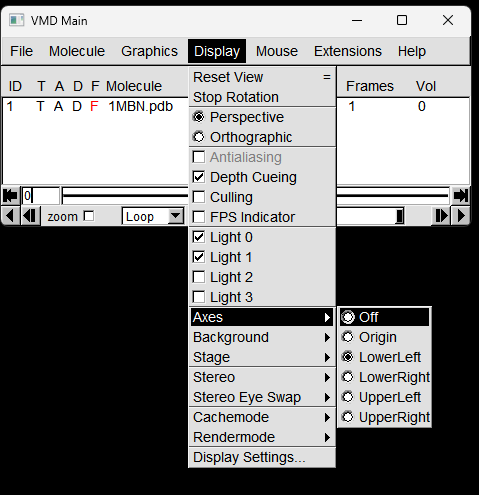
VMD 软件界面如下图所示,主要由3部分组成,如下图所示。
第一部分是VMD的显示窗口(OpenGL Display); 第二部分是VMD的主窗口(VMD Main) 第三部分是VMD的命令窗口;
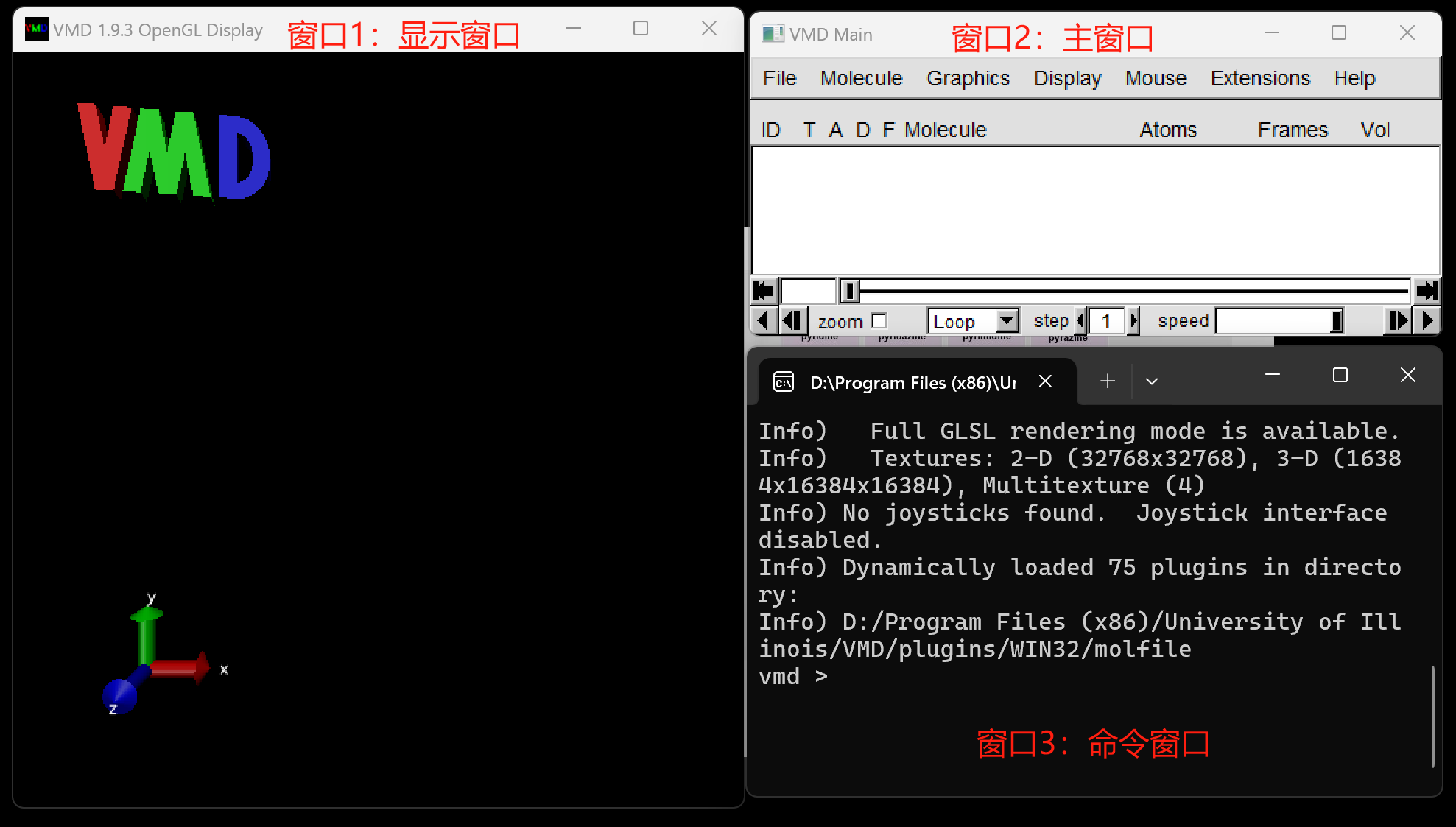
VMD main窗口¶

- ID: 每个分子都有唯一的ID
- T(Top): 只有一个分子是TOP分子,顶层分子
- A(active): 分子处于激活状态,参与动画播放
- D(Display): 是否显示该分子
- F(fixed): 当前分子是否固定不动,鼠标拖拽旋转时,分子不动。
- Molecule: 分子名称,双击可修改
- Vol:电子密度图、静电势分布、占有率图等网格数据
载入单个蛋白结构(PDB)¶
以肌红蛋白(PDB ID: 1MBN ) 为例进行介绍。 文件:https://files.rcsb.org/download/1MBN.pdb
在主窗口(VMD main)中点击FILE -> New Molecule
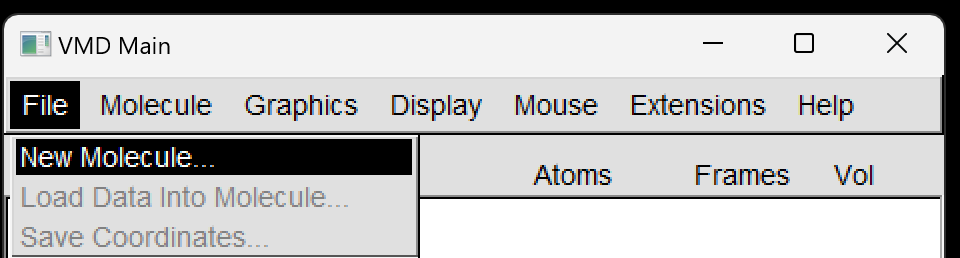
然后点击Browse 找到需要打开的PDB文件,再点击Load,就可以载入蛋白。
在VMD Display 窗口中,我们可以看到肌红蛋白,如下图所示:
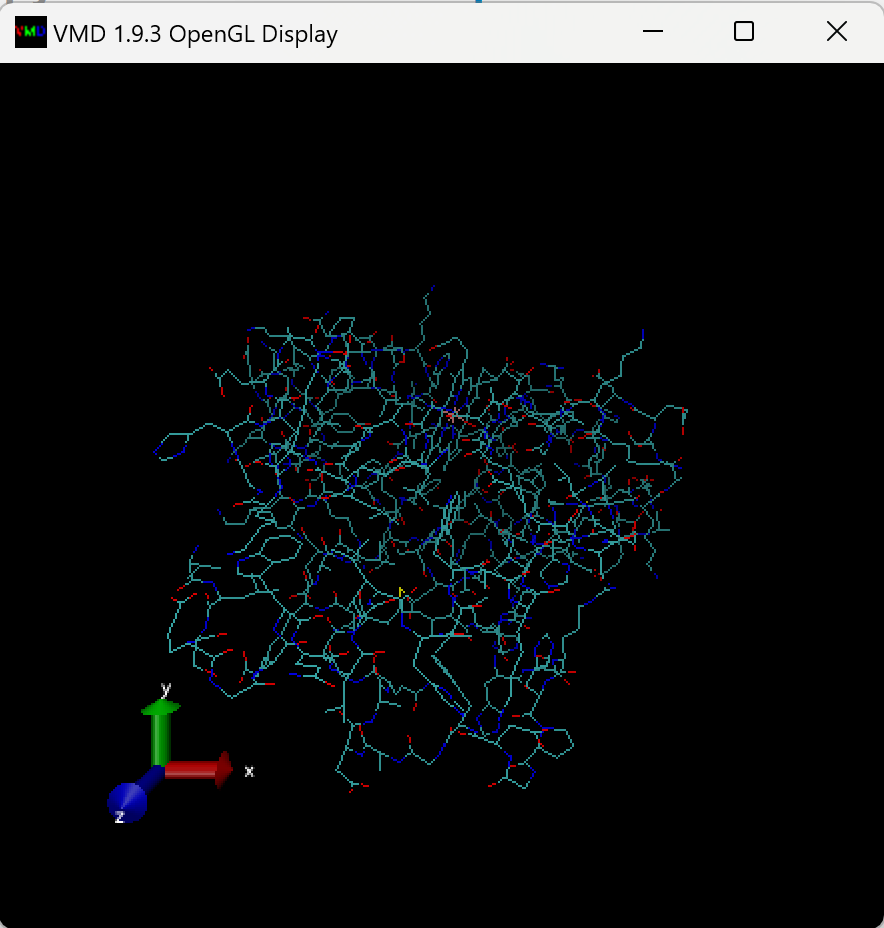
默认是以lines的形式显示的蛋白。
肌红蛋白的cartoonb表现形式绘制¶
肌红蛋白(PDB ID: 1MBN ) 含有辅因子铁血红素(Ferroheme) 。 蛋白以PyMol中的cartoon形式进行展示,基于二级结构自动着色。 血红素以stick的形式进行展示。
注解
PyMol 和 VMD 都有PhotoShop层Layer的概念。 其中VMD 一切都是层Layer。 理解这个概念非常重要。
step1. 在VMD MAIN窗口的菜单点击Graphics -> Representations… 打开Graphical Represetations窗口,如下图所示。
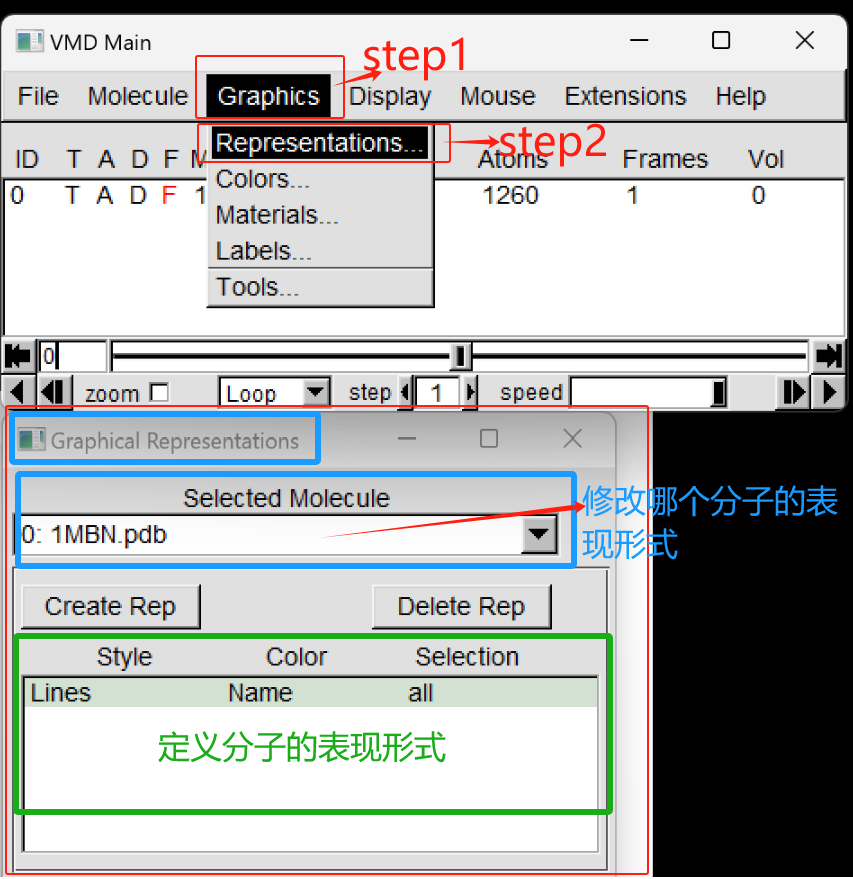
从上图,我们可以看到我们处理的分子是1MBN,只有lines一种表现形式。
注解
Drawing Method 方法有很多种,先不一一介绍。 后面会详细介绍每一种方法对应的形式。
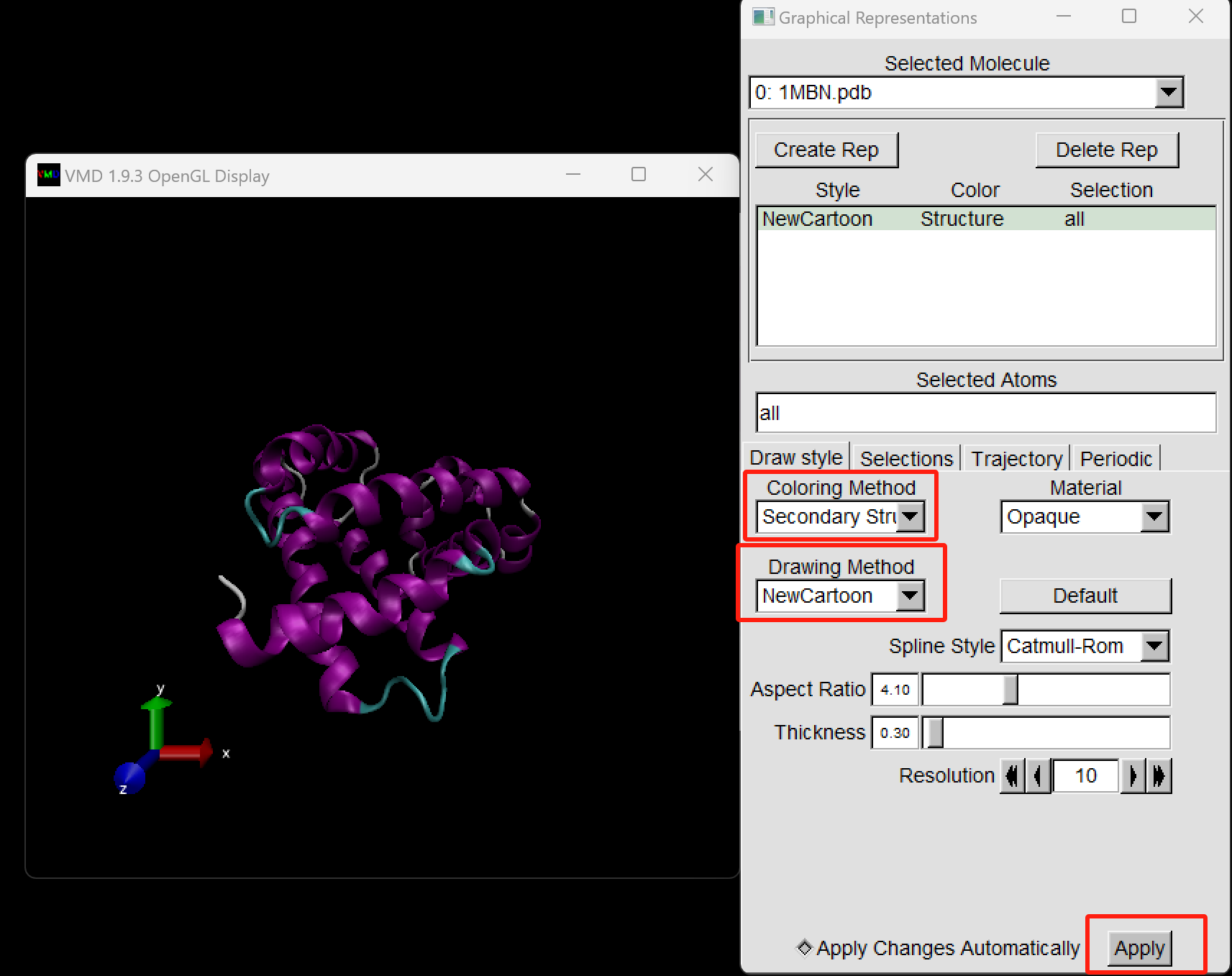
step2. 修改蛋白为PyMol的cartoon形式。 首先我们Drawing Method方法从 lines 修改为 NewCartoon, 以及把coloring method方法从 Name 修改为Secondary Structure, 最后点击Apply, 如下图所示,我们可以看到蛋白结构变为PyMol的Cartoon形式了。
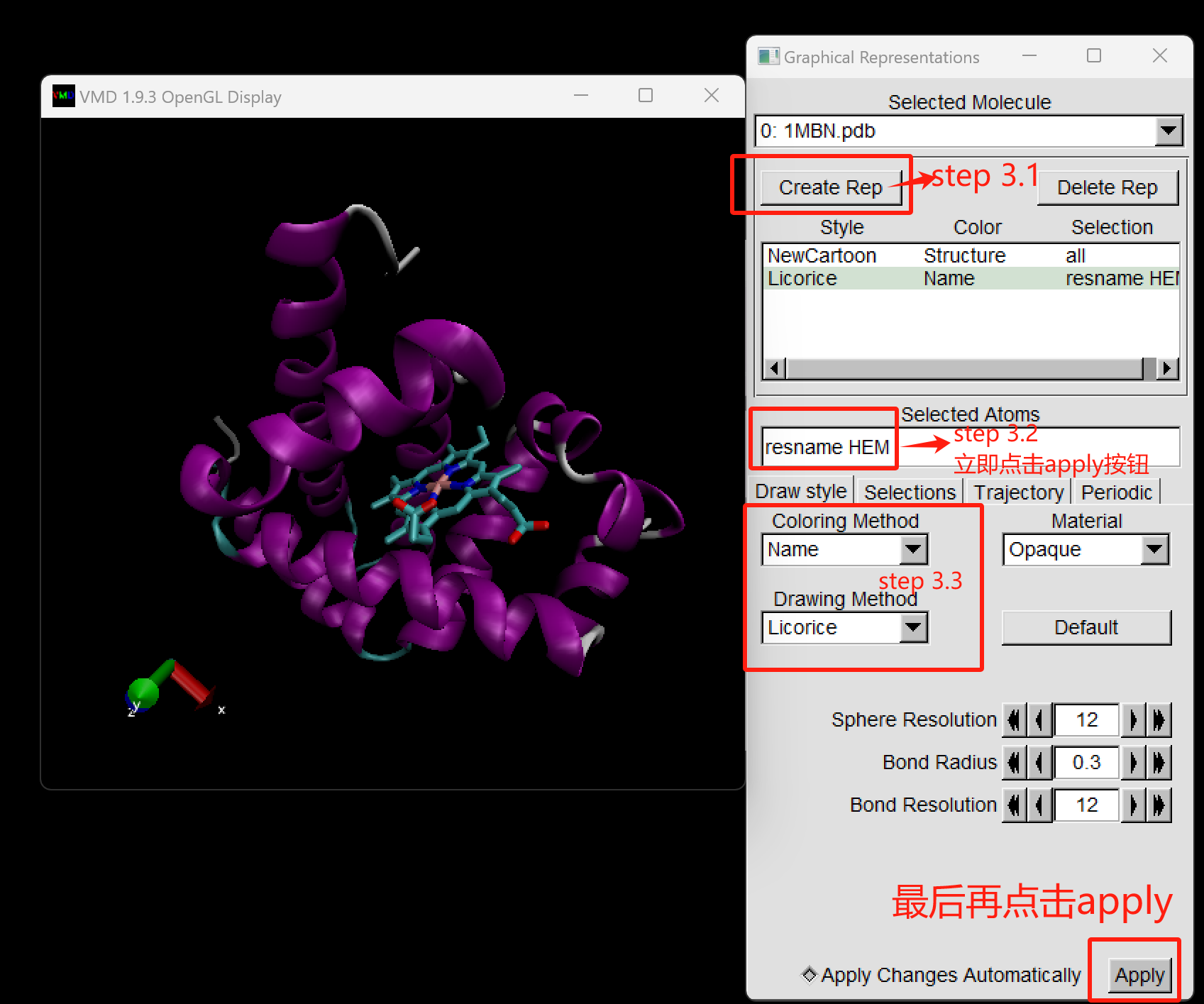
step3. 修改铁血红素为stick形式。
注解
VMD的显示窗口,只有显示功能,没有其他功能。 选择配体的功能,都是在Graphical Represetations窗口中完成。
从 1MBN 网站上我们知道辅因子铁血红素的 残基名称是HEM.
step 3.1 在Graphical Represetations窗口中,点击Create Rep,给这个分子创建了一个层, 这个层后续用来显示HEM。
step 3.2 在selectd atoms 框中输入 resname HEM 并点击apply;
注解
确定好原子后,一定要立即点击apply使生效。
step 3.3 首先我们Drawing Method方法修改为 Licorice, 以及把coloring method方法修改为 Name,bing点击apply;如下图所示。
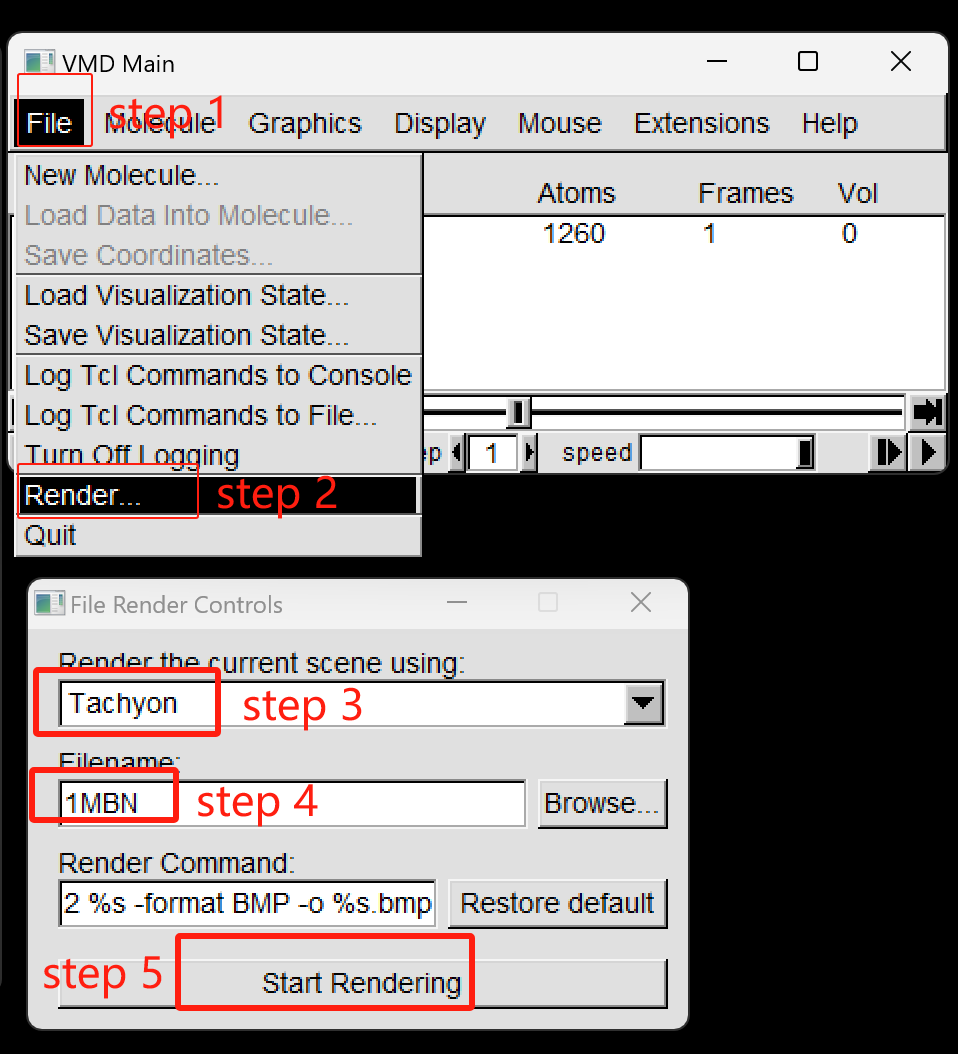
渲染和导出图片(Tachyon)¶
VMD 内置了多种渲染图片的算法,其中Tachyon是最常用的算法。
首先在VMD命令行窗口查看和修改当前的工作路径。
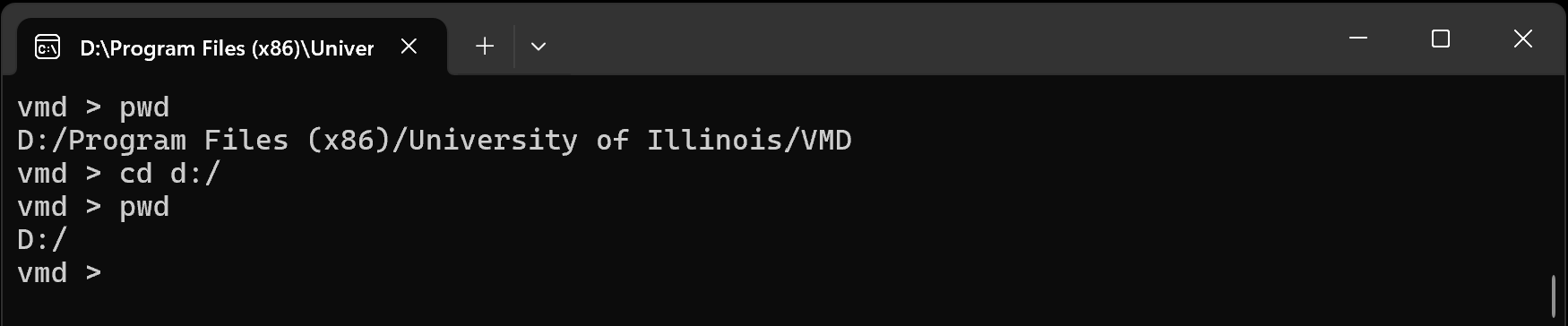
我们把工作路径从VMD的安装目录 D:/Program Files (x86)/University of Illinois/VMD 切换到D盘根目录 D:/。
注解
pwd(print working directory”)命令会输出当前的工作目录。
图片默认会导出到该目录下面。
可以通过cd 命令切换工作目录。
然后在VMD main 窗口点击 File -> Render 打开 File Render Controls窗口。 接着选择渲染算法 Tachyon, 设置导出的图片名称1MBN,最后点击Start Rendering。
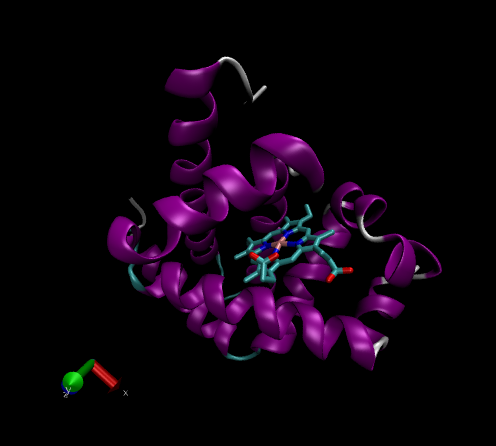
在D盘的根目录下有 D:/1MBN.bmp图片,如下图所示。
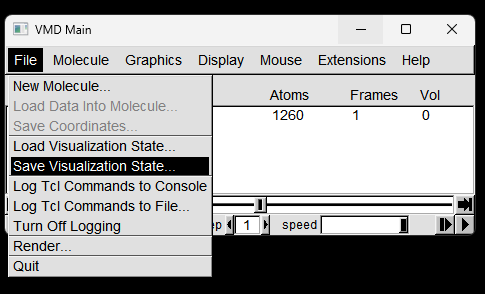
保存VMD的状态¶
保存VMD的状态,生成vmd 文件,类似PyMol的pml 文件。
在VMD main 窗口点击File -> Save Visualization State…
保存文件为 1MBN.vmd
1MBN.vmd 。
注解
xxx.vmd 文件不包含分子本身的数据。 是VMD命令的文本文件。因此要确保当前目录下面有xxx.vmd 需要的所有分子文件。
如果共享vmd文件给其他人,也要同时共享分子文件, 同时用文本打开vmd文件,注意其中的路径。
导出分子的坐标¶
- 在VMD MAIN的分子列表窗口选择感兴趣的分子;背景为绿色代表分子被选中。
2. 鼠标右击感兴趣的分子,选择Save Coordinates… 或者在VMD MAIN的窗口,点击File->Save Coordinates…
如下图所示:
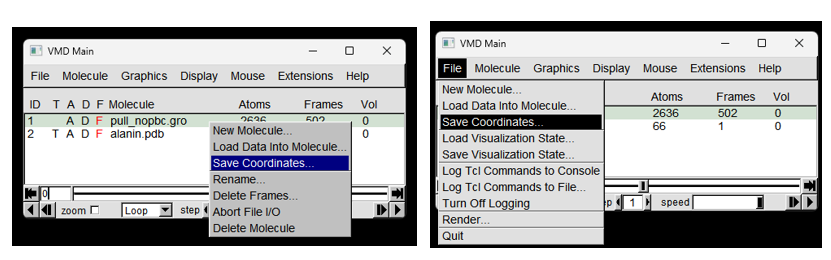
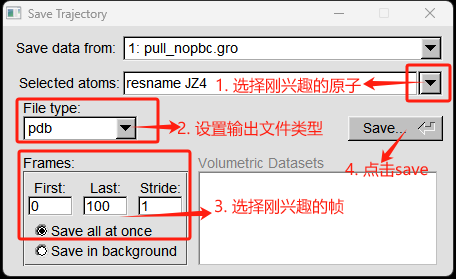
- 点击下拉框列表,选择感兴趣的原子;或者自定义感兴趣的原子。
注解
下拉框和Graphic Representation中的内容一致
- 设置感兴趣的帧数。
5. 点击save 如下图所示:
分子的显示方式¶
Drawing Method: 1. 蛋白的显示方式:
1. Cartoon (卡通模型):用螺旋和带状结构表示蛋白质的二级结构,适合展示蛋白质的α螺旋、β折叠和环结构,直观显示整体折叠方式。
2. NewRibbon (新带状模型):类似于 Cartoon,但更适合展示更详细的二级结构,尤其是当你需要对蛋白质结构的走向有更清晰的了解时。
3. Surface (表面模型):显示蛋白质的分子表面结构,适合分析蛋白质与其他分子的相互作用,尤其是识别活性位点和相互作用区域。
4. Licorice (甘草模型):有时也用于蛋白质的小部分,例如显示活性位点的氨基酸或分析配体结合。
- 小分子的显示方式:
1. CPK (球棒模型):用于突出小分子原子的大小和键的分布。球体表示原子,棒表示键,适合用于查看分子的整体形态和相互作用。
2. Licorice (甘草模型):显示键之间的连接关系,适合分析小分子的键和原子的相对位置。
3. Lines (线条模型):用简单的线条表示原子间的键,适用于展示小分子的整体结构,并减少视觉复杂度。
4. VDW (范德华表面):展示小分子的原子范德华力半径,适合用来研究分子的表面与其他分子或环境的相互作用。
原子选择(selected atoms)¶
修改背景颜色¶
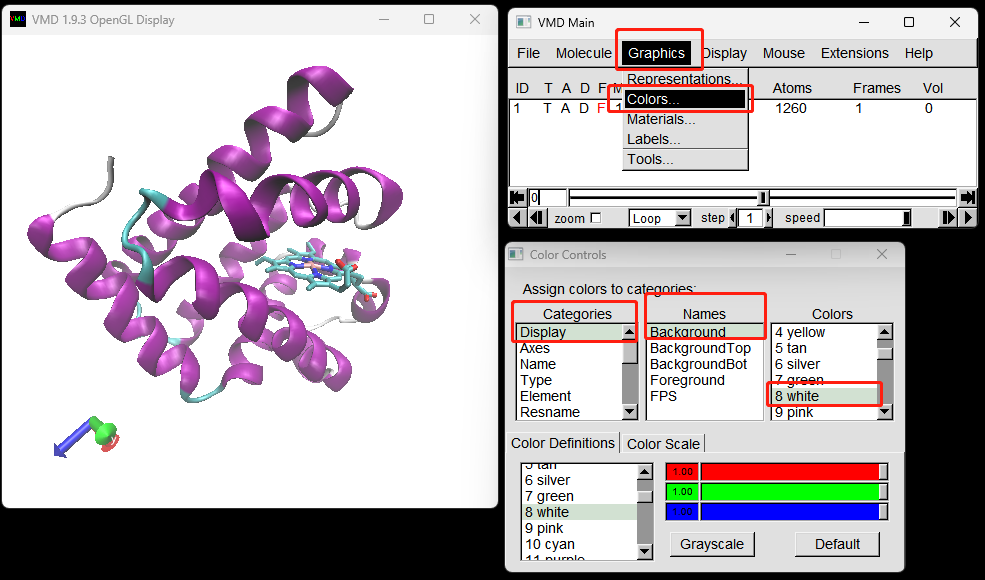
VMD默认背景颜色是黑色的,发表文章图片背景通常是白色。
黑色背景省电,白色背景省墨。
在VMD main 窗口点击 Graphics -> Colors… 打开 color controls 窗口。 然后在 color controls 窗口进行设置, Categories 中选择Display, Names 中选择Backgriund, Colors 中选择8White 。
显示蛋白表面(Surf)¶
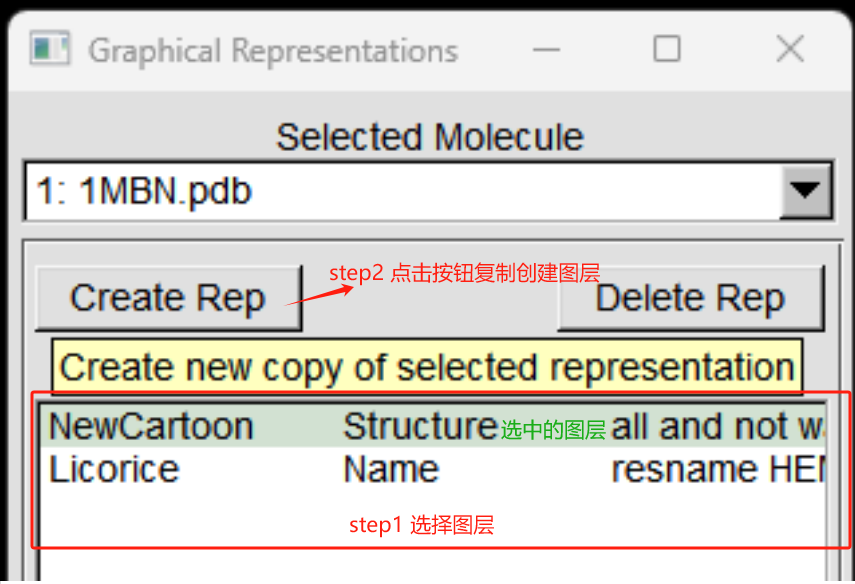
在VMD MAIN窗口的菜单点击Graphics -> Representations…
step 1 选择图层创建复制新图层。
注解
Create Rep 代表的是Create new copy of selected representation。
因此我们需要先选择合适的图层,然后再点击Create Rep按钮。
step 2 对新创建的图层进行设置,修改Drawing Method为Surf; Coloring Method 为Color ID 和8; Material 为Transparent 。如下图所示,
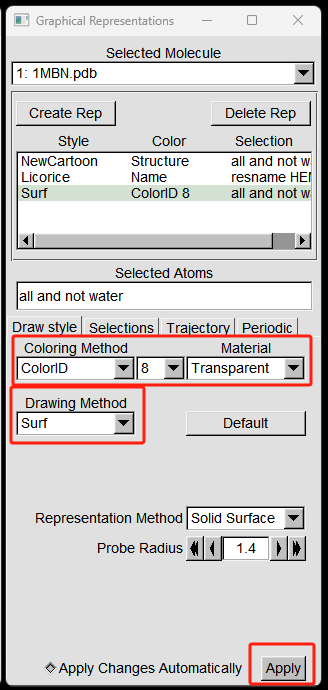
生成的蛋白表面如下图所示:
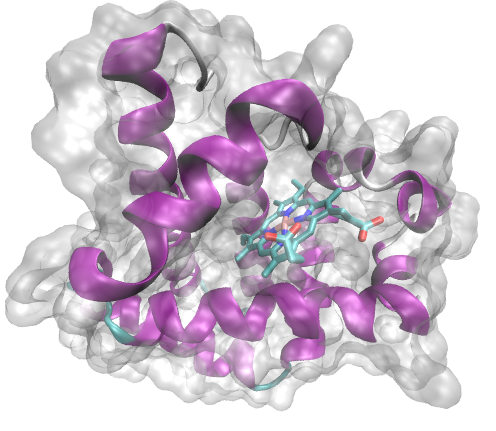
创建Label¶
为了标注图片中的各个对象是什么,可以通过Label进行创建。
step1. 首先将鼠标从默认的Rotate Mode切换到Label模式, 在VMD Main窗口,点击 Mouse -> Label -> Atom1,如下图所示。
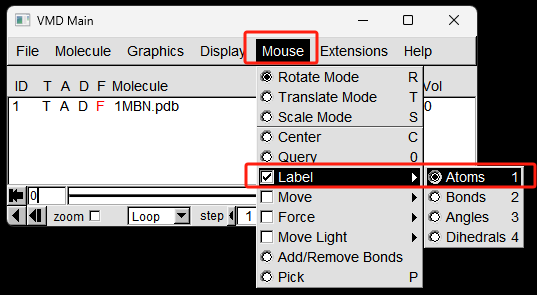
切换成功后,鼠标放到OpenGL Display窗口会变成十字架。
step2. 在openGL 窗口点选感兴趣的原子,比如点击辅因子HEM中心原子;
step3. 修改Label的颜色
默认Label的颜色是绿色,这里我将颜色修改为粉色(Pink)。
在VMD Main窗口中点击 Graphics -> Colors… , 打开Color Controls窗口。 Categories 中选择 Labels, Names中选择Atoms, Colors 从默认的7 green 修改为 9pink, 如下图所示。
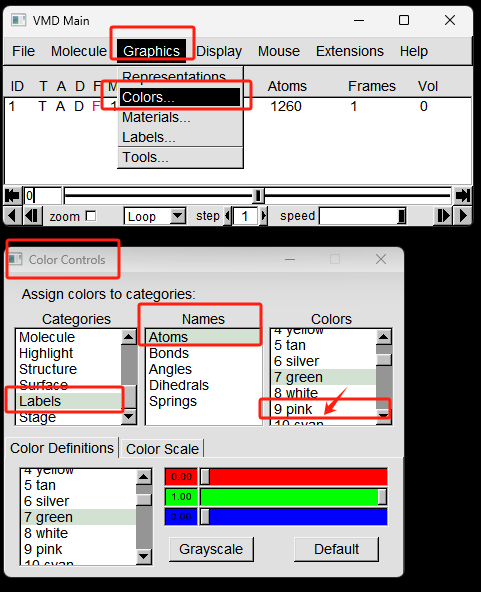
step4. 修改Label内容和移动Label到合适位置
在VMD Main窗口中点击 Graphics -> Labels… , 打开Labels窗口。
1. 选择Label的类型atoms;
2. 点选需要修改的label, 选中后label的背景颜色变成绿色;
3. 点选Properties标签,
4. 在Format文本框中修改Label的内容为Fe
5. 移动Label的位置,使得Label 更加清晰。
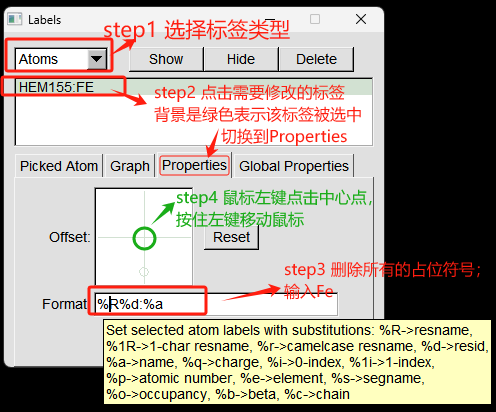
step5. 修改Label的大小和粗细。
在Labels窗口,
1. 点选Properties标签,
2. 在Format文本框中修改Label的内容为Fe
3. 移动Label的位置,使得Label 更加清晰。
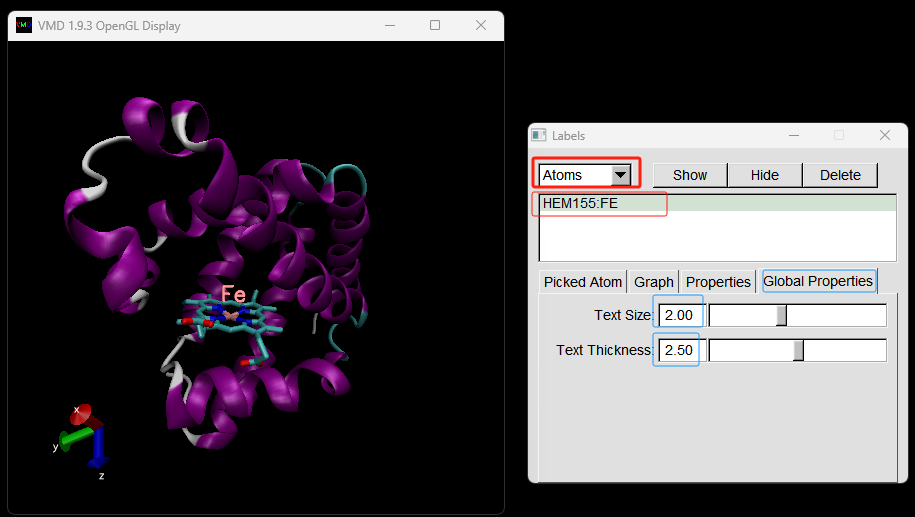
注解
Mouse 在Rotate Mode + Label模式下, 按住鼠标左键移动进行旋转分子; 滚动鼠标中键进行缩放操作。 熟练使用鼠标可以点击任意想要点击的原子。
点击一下原子,会创建Label, 再点击一次该原子会删除Label 。
测量距离(bond)¶
测量任意两个原子的距离,将鼠标模式切换到rotate 模式+Label bond模式。
方法1:在VMD Main 窗口,点击Mouse->Rotate 和Mouse->label->bond。
方法2:在Display 窗口,点击R(代表Rotate) 和 2(代表bond)。
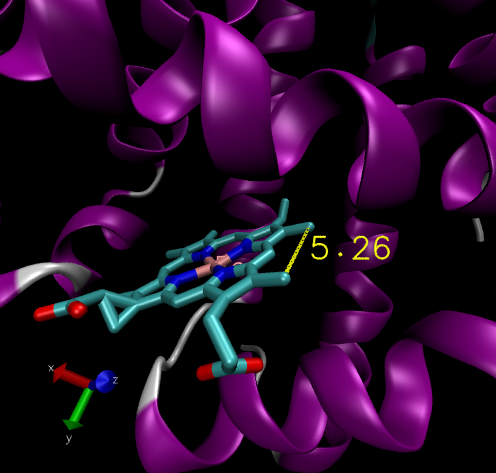
调整视图,然后鼠标依次点击2个原子,完成距离的测量, 会自动给2个原子添加Label,以及距离添加Label,如下图所示。
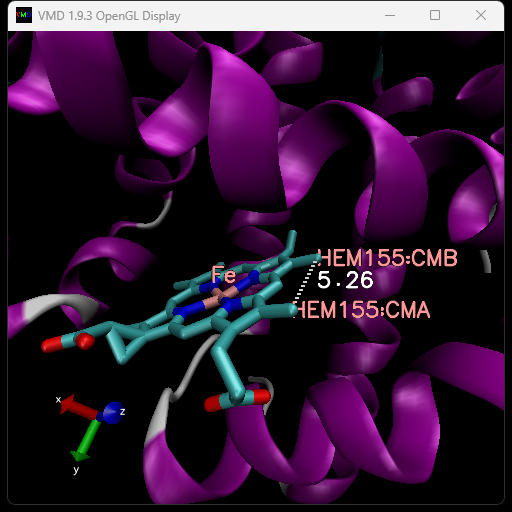
如上图所示,CMA和CMB两个原子的距离是5.26Å 。
删除原子的Label:
在VMD Main 窗口,点击Graphics->Labels 打开Label窗口。 首先选择 Atoms,然后按住shift 或者 Ctrl 选择所有你不要的Label,最后点击删除,如下图所示。
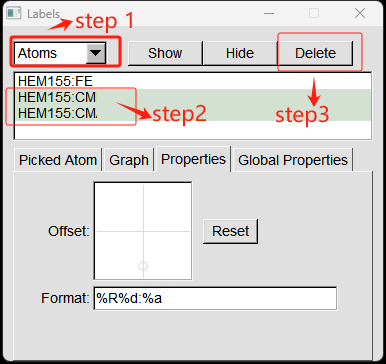
修改距离的颜色:
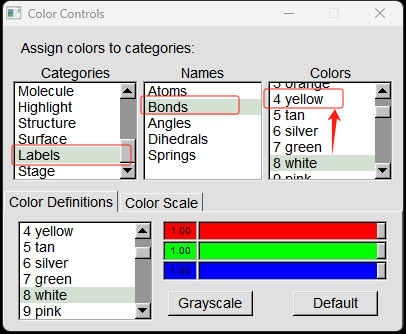
在VMD Main 窗口,点击Graphics->Colors 打开Color Controls窗口。
如下图所示,依次点击Labels-> bond-> 4yellow。
输出效果图:
注解
在VMD中测量等同于Label。
测量角度(Angle)¶
测量任意三个原子的角度,将鼠标模式切换到rotate 模式+Label Angle模式。
方法1:在VMD Main 窗口,点击Mouse->Rotate 和Mouse->label->Angle。
方法2:在Display 窗口,点击R(代表Rotate) 和 3(代表angle)。
调整视图,然后鼠标依次点击3个原子,完成角度的测量。
测量二面角(Dihedral)¶
测量任意两个原子的距离,将鼠标模式切换到rotate 模式+Label Dihedrals模式。
方法1:在VMD Main 窗口,点击Mouse->Rotate 和Mouse->label->Dihedrals。
方法2:在Display 窗口,点击R(代表Rotate) 和 4(代表Dihedrals)。
调整视图,然后鼠标依次点击4个原子,完成二面角的测量。
鼠标模式¶
熟练切换鼠标模式,可以调整获得任意你想要的视图和信息。
在VMD Main 窗口,点击Mouse, 一共支持3个基本模式和8个拓展模式,如下图所示。
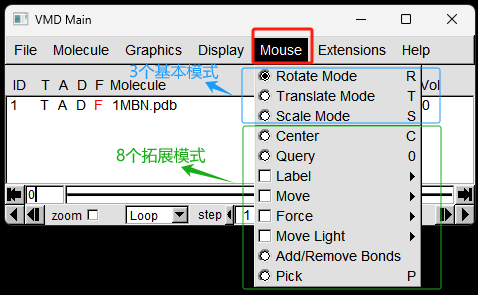
3个基本模式:
1. 旋转模式(R),默认模式;在VMD Main->Mouse->选择Rotate Mode; 或者在Display窗口,按R或者r 进行切换。
2. 平移模式 (T);在VMD Main->Mouse->选择Translate Mode; 或者在Display窗口,按T或者t 进行切换。
3. 缩放模式(S);在VMD Main->Mouse->选择Scale Mode; 或者在Display窗口,按S或者s 进行切换。
旋转模式下(R):
1. 按住鼠标左键可以上下左右自由旋转;
2. 滚动鼠标中键可以进行缩放;
3. 按住鼠标右键可以沿着屏幕法线方向旋转;
平移模式下(T):
1. 按住鼠标左键可以上下左右自由移动;
2. 滚动鼠标中键可以进行缩放;
3. 按住鼠标右键可以前后移动,较少使用;
缩放模式下(S):
1. 按住鼠标左键可以缩放;
2. 滚动鼠标中键可以缩放;
3. 按住鼠标右键可以缩放;
8个拓展模式:
注解
拓展模式不可以单独使用;一定是和基础模式搭配使用。
- 拓展模式-Label
比如我在R模式下,对原子进行Label,
在Display窗口,先按R/r, 确认进入到旋转模式, 调整视图,然后按1进行原子label 模式,点选原子。
注解
所有的鼠标模式,都可以通过快捷键切换。 入门:记住3个常用的快捷键; 掌握:记住9个快捷键; 精通:记住所有的快捷键;
渲染模式RenderMode¶
VMD支持3种渲染模式:
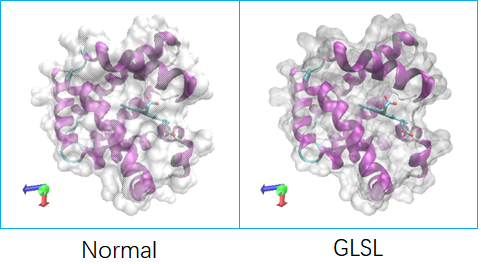
- Normal: 这是 VMD 的默认渲染模式,使用基本的 OpenGL 图形指令进行渲染。能快速显示分子结构和表面。
- GLSL: 能提供更精细的视觉效果,比如更真实的光影、材质、和反射效果。适合生成高质量的分子可视化图像。
- Acrobat3D: 用于生成与 Adobe Acrobat 3D 兼容的图形输出。这种格式允许将 3D 模型嵌入到 PDF 文件中,便于分享和展示。
通过在VMD Main 窗口中,点击Diplay -> Rendermode 进行选择不同的渲染模式。
注解
正常情况,选择Normalm模式。
发表文章或者显示蛋白表面,切换到GLSL模式,更加清晰。
Graphical Representations中的Atom selecton¶
蛋白的表现形式(Representations)¶
载入动力学轨迹(gromacs)¶
VMD支持多种格式的分子动力学轨迹格式,这里以gromacsamberschrodiger为列进行介绍。
载入gromacs格式的轨迹,需要结构文件(gro)和轨迹文件(xtc)文件。
这里我以T4蛋白的拉伸动力学轨迹为例进行介绍。
下载`T4轨迹 <https://pan.baidu.com/s/1vBAvMxRZ6h-kONU-ccoUYw?pwd=68u5>`_文件:
https://pan.baidu.com/s/1vBAvMxRZ6h-kONU-ccoUYw?pwd=68u5
一共包含2个文件:pull.gro 和 pull.xtc 。
首先在VMD main 窗口,点击File -> New Molecule…, 打开Molecule File Browser窗口。然后点击Browse 找到pull.gro文件。最后点击load。
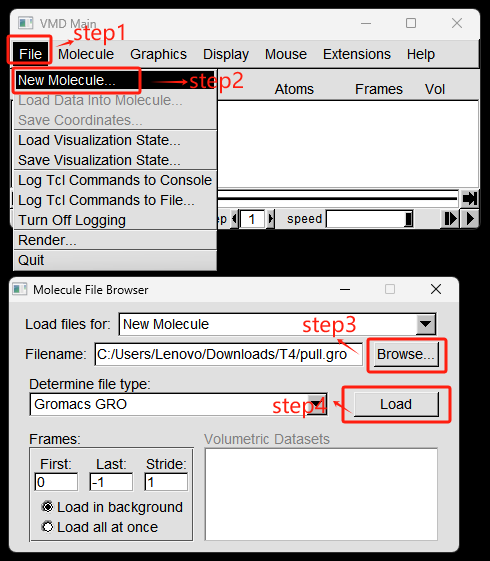
接着在Molecule File Browser窗口。 确认和选择load files for:对应0:pull.gro名称; 点击 Browse 找到pull.xtc文件。 最后点击load。
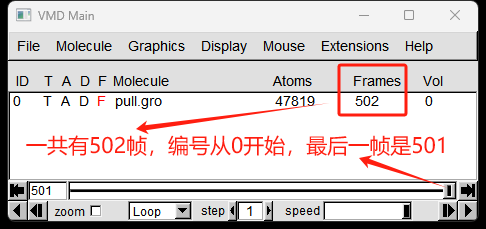
加载完毕,一共有502帧。
注解
图形界面载入大轨迹速度比较慢,建议通过命令行载入大轨迹。 比如50000帧的轨迹,命令行( vmd first_frame.gro md_100ps_nojump_fit.xtc ) 载入,不到1分钟就可以载入。 图形界面载入需要 20 分钟。
载入动力学轨迹(amber)¶
注意VMD只能载入文本文件,如果是二进制文件,需要先转换成文本文件。
载入xxx.prmtop 和 equil.rst 文件。 默认 rst文件是二进制文件,需要通过cpptraj命令转换成rst7文件。
cpptraj -p xxx.prmtop -y equil.rst -x equil.rst7
方法1:终端命令行中直接输入 vmd xxx.prmtop equil.rst7
方法2:
首先在VMD main 窗口,点击File -> New Molecule…, 打开Molecule File Browser窗口。然后点击Browse 找到xxx.prmtop文件。最后点击load。
接着在Molecule File Browser窗口。点击 Browse 找到equil.rst7文件。最后点击load。
载入schrodinger格式的轨迹
配置videomach¶
VideoMach 是一款视频编辑和转换工具,VMD 制作动画默认用的是videomach。
如何配置videomach 变量,使得VMD 能够自动找到videomach。
首先下载并安装 VideoMach 。
我的安装路径是 C:Program Files (x86)VideoMachvideomach.exe 。
基于轨迹制作动画¶
素材: T4蛋白的拉伸动力学模拟
包含3个文件: 1. pull_nopbc.vmd; 2.pull_nopbc.gro; 3.pull_nopbc.xtc
通过网盘分享的文件:T4_pull_nopbc.zip
链接: https://pan.baidu.com/s/1CQcU8xwY7pvjVNu0LBTaww?pwd=dx22 提取码: dx22
注解
将3个文件放到VMD的工作目录中, 这样可以通过File->load visual state 加载VMD 文件;
在制作动画之前,确保安装并 配置好videomach 。
如下图所示,点击Extensions->Visualization->Movie Maker,打开动画制作窗口。
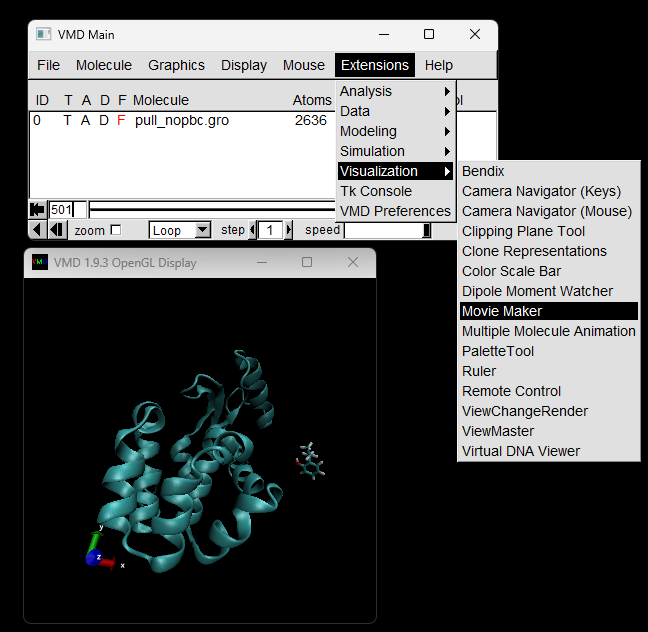
在动画制作窗口中,首先 Moving settings中选择trajectory, 然后依次设置输出目录,动画名称;最后点击制作动画按钮。
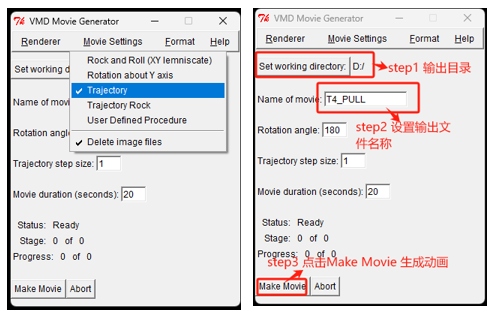
输出动画:

动力学轨迹中距离变化的可视化¶
首先按照这个`方法 <http://vmd.chenzhaoqiang.com/intro/startManual.html#bond>`_ 测量感兴趣的距离,
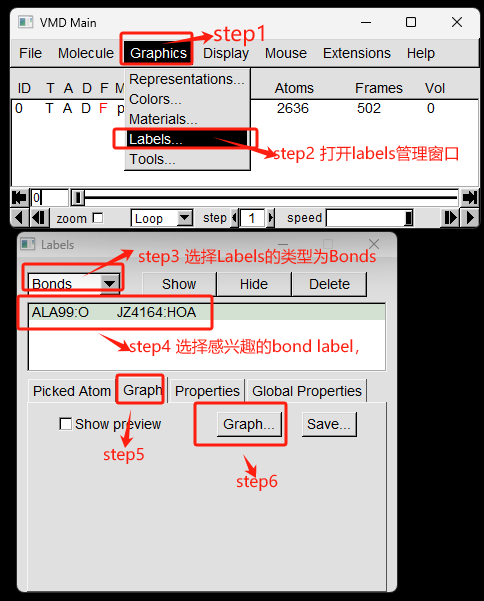
这里我们对99号氨基酸ALA和配体JZ4形成的氢键距离比较感兴趣,我们首先利用鼠标的bond模式,测量距离。 然后点击Graphics->Labels 打开labels管理窗口, 再在labels管理窗口下,依次选择Labels的类型为Bonds, 选择感兴趣的bond label, 点击Graph标签,最后点击Graph…按钮,如下图所示:
输出:
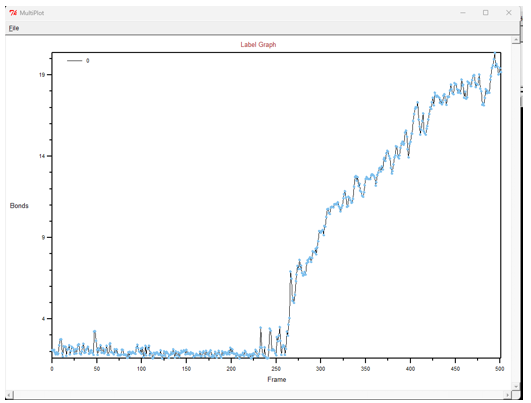
从这里我们可以看出小分子大约在270左右开始从口袋中解离。
注解
VMD 适合快速查看结果。 发表文章建议用python重新绘图。
VMD配置文件.vmdrc和vmd.rc¶
The .vmdrc and vmd.rc files
在windows中总的配置文件位于VMD的安装路径中。 比如我的vmd.rc 位于 D:Program Files (x86)University of IllinoisVMDvmd.rc
打开vmd.rc 文件,
# mol default selection {name CA}
# mol default material {Transparent}